癌症基因组的突变避免了转录因子识别的位点
基因组突变在癌细胞中比在正常细胞中发生的频率高得多。突变可能不可预测地破坏各种基因组元件并影响重要的细胞功能。直到最近,在大多数情况下,只有癌症基因组的编码区域可用于外显子组测序项目的研究。因此,与基因组的其他部分相比,这些区域的突变得到了更好的研究。随着大规模癌症全基因组测序的最新进展,这种情况发生了变化,这些测序获得了全基因组范围内的突变信息,包括非编码区。

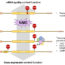
图1所示。位于AP2A(上图)和ESR1(下图)转录因子结合位点内及附近的乳腺癌突变的相对频率。洗牌序列和随机取样的基因组片段作为对照。体细胞突变在AP2A结合模式的TGA环境中富集,在ESR1结合模式的TCA环境中耗尽。
非编码基因组的一个显著部分是由调控片段组成的,它控制着不同细胞类型中基因的活性。这些片段的核苷酸序列,启动子和增强子,包含转录因子识别的多种模式,这些转录因子是帮助启动RNA合成机制或调节其效率的特定蛋白质。
基因组突变可以破坏转录因子识别的DNA序列模式,改变DNA-蛋白质结合亲和力,进而改变靶基因的活性。这可以改善或干扰突变细胞系的存活。因此,携带特定突变的细胞数量取决于突变位置。通过同时研究许多细胞样本和DNA位点,有可能观察到哪些DNA模式会积累比偶然预期更多的突变。这对应于特定序列背景下突变的正选择,这种类型的选择已经在早期的出版物中报道过。另一方面,负选择,即特定序列背景积累的突变比偶然预期的要少,仍然难以捉摸。
我们对之前在不同癌症类型的样本中发现的数百万个非编码突变进行了计算分析。我们发现,人类癌细胞的基因组在某些情况下积累的突变比预期的要少,而在这些情况下,突变改变了转录因子识别的模式。特别是,对于TCA三核苷酸(反向DNA链上的TGA)内高度突变的胞嘧啶,在某些癌症类型(例如乳腺癌)中,被APOBEC3脱氨酶攻击并转化为TTA的胞嘧啶,情况也是如此。在某些情况下,如果这些三核苷酸位于代表所选转录因子结合位点的较大模式中,则比预期更频繁地发生突变(图1,顶部面板)。这反映了积极选择。同时,对于其他模式,突变频率显著降低(图1,底部面板),突出了负选择。
考虑到各种情况下的所有突变,我们发现与各种转录因子结合相对应的多个模式家族的突变频率明显低于预期。这种选择压力似乎可以保护癌细胞免受特定调节回路的重新布线。值得注意的是,类似的效应早前在正常基因组的多态性位置被观察到。
对具有抗突变结合基序的转录因子的进一步分析可以确定对不同人类癌症的生存和进展至关重要的特定调控途径。
Ivan Kulakovskiy, Ilya Vorontsov, Vsevolod Makeev
瓦维洛夫普通遗传学研究所,俄罗斯,莫斯科
恩格尔哈特分子生物学研究所,俄罗斯,莫斯科
出版
负选择在人类癌症中维持转录因子结合基序。
罗龙索夫,李建军,李建军,李建军,李建军,李建军,李建军
BMC Genomics 2016年6月23日












留言回复
你一定是登录发表评论。