活细胞的超分辨率:细胞分裂机制的分子组织可视化
我们着手阐明细胞动力学装置蛋白质的分子组织,这是理解细胞分裂机制的重要一步。细胞分裂在单细胞模式生物裂变酵母中得到了最好的理解。在活的分裂酵母细胞中,荧光共聚焦显微镜在鉴定参与细胞质分裂的蛋白质、计算这些蛋白质的数量和描绘导致细胞分裂的事件的时间线方面发挥了关键作用。在这种模式生物中,细胞动力学收缩环通过节点的合并组装,蛋白质组装包含多个结构蛋白和调节蛋白。这些蛋白质是在节点中共定位还是每个蛋白质在节点的不同子集中分布?蛋白质是被组织成产生力的基本单位还是随机分布在细胞动力学装置内?节是裂变酵母收缩装置的基本功能单位吗?在收缩环收缩过程中,有丝分裂蛋白是否保持较高的有序结构?

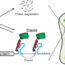
图1所示。有丝分裂结蛋白的分子模型。(A)分裂酵母细胞的横截面图,通过聚焦于细胞表面或中间获得物体的不同视图,(下)FPALM在正面和侧面视图中节点的重建图像。(B)已知节点蛋白相互作用示意图(C)细胞质分裂节点中6种蛋白的位置示意图(左图)和细胞顶部的正面图(右图)。
我们用活分裂酵母细胞的高速荧光光激活定位显微镜(FPALM)解决了这些问题,并获得了~35 nm分辨率的图像,比共聚焦荧光显微镜的分辨率提高了大约10倍。我们用光激活荧光蛋白mEos3.2标记基因组中的细胞分裂蛋白。我们将这些标签放置在6种高度保守的细胞分裂蛋白的8个不同位点上:苯胺同源蛋白Mid1p、含有F-BAR结构域的蛋白Cdc15p、formin Cdc12p、IQGAP蛋白Rng2p、肌球蛋白ii重链Myo2p及其调控轻链Rlc1p。我们对表达单个标记蛋白或对标记蛋白的活细胞进行成像。我们通过聚焦于细胞的表面或中间部分来获得节点的正面或侧面视图,以提供两个视角(图1A)。我们对单个光激活的mEos3.2分子进行了成像,并在空间和时间上精确定位了每个单分子发射源。我们建立了关键的分析工具来测量蛋白质簇的尺寸和形状,计算每个蛋白质簇的定位发射器的数量,并利用我们数据集中获得的数千个单分子定位的定量潜力。
8个不同mEos3.2标签在节点中的不同分布表明节点是包含所有6种蛋白质的离散结构。我们提出了一个蛋白质在节点中排列的三维模型,该模型受每个蛋白质的数量、电子显微镜和x射线晶体学中蛋白质的形状和大小、mEos3.2定位相对于细胞边缘的位置以及蛋白质之间已建立的相互作用的限制(图1B,C)。
在我们的模型中,anillin/Mid1p位于靠近质膜的淋巴结核心。IQGAP/Rng2p将Mid1p连接到Myo2尾部的末端。F-BAR蛋白Cdc15p连接Mid1p和形成蛋白Cdc12p。将Myo2的尾巴固定在核心蛋白上,产生了一个运动单元,在几何上相当于动物和变形虫的双极性肌球蛋白- ii细丝,但具有径向对称。Myo2的这种花束状排列使头部呈扇形伸展到细胞质中,在那里它们可以在收缩环形成和收缩过程中捕获肌动蛋白丝。
FPALM提供的更高分辨率还揭示了节点在收缩环中持续存在,当环收缩时,节点围绕环的圆周双向移动。
利用FPALM对活细胞进行定量成像是在纳米水平上研究动态细胞系统的有力方法。了解多蛋白复合体中蛋白质的空间组织结构有助于阐明每个组成蛋白的作用和生物学功能。细胞动力学蛋白在分裂酵母中被组织成离散的功能单位的发现,为其他生物体中细胞动力学环的组织提供了一些线索。
托马斯·d·波拉德,卡罗琳·拉普兰特
耶鲁大学分子细胞与发育生物系,美国康涅狄格州纽黑文
耶鲁大学细胞生物系,美国康涅狄格州纽黑文
耶鲁大学分子生物物理与生物化学系,美国康涅狄格州纽黑文
美国北卡罗来纳州罗利市北卡罗莱纳州立大学分子生物医学科学系
出版
活裂变酵母细胞分裂节点和收缩环的超分辨荧光显微镜下的分子组织。
黄峰,张建军,张建军,张建军
中国科学院学报2016年10月4日












留言回复
你一定是登录发表评论。