改进了可达表面积的预测
蛋白质由由肽键连接的氨基酸残基线性链组成。蛋白质通过其三维(3D)形状或结构来执行一系列功能,形状可以从其一维(1D)氨基酸序列中确定。给定一维序列信息,就可以确定蛋白质的三维形状,然而,这种从一维到三维的映射是一项具有挑战性的任务。预测可达表面积(ASA)可以帮助确定蛋白质的形状。
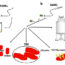
ASA是指生物分子(残基)的表面积,在探测该分子表面时,球形溶剂可以接近该分子(见图1)。大多数蛋白质分子具有疏水核心,溶剂无法接近该核心,并且具有与环境接触的极性表面。因此,氨基酸残基的ASA决定了相互作用窗格,最终在蛋白质结合机制、结构和功能中发挥重要作用。
残留物可以分为暴露的或埋藏的,也可以用一个真实的值来量化,给出一个更实际的连续变化的表面积。我们试图直接从蛋白质的氨基酸序列中预测蛋白质残基的真实ASA值。
我们提出了一种新的基于序列的ASA统计预测因子,即REGAd3.p.为此,我们构建了一个包含1299个蛋白质序列的基准数据集SSD1299,用于训练和独立测试预测模型。我们生成了一套完整的55个特征,可以反映蛋白质序列与其残基的asa之间的内在相关性。作为这些特征生成步骤的一部分,我们分别构建了自己的蛋白质二级结构(螺旋、β和线圈)预测模型,并将其输出用于ASA预测。采用正则化精确回归技术,结合遗传算法优化权重,建立ASA预测模型,计算ASA预测值。具体来说,我们将这些特征提供给算法,以便从训练数据集中了解ASA的适当特征,然后评估其在测试数据集中的性能。
我们根据平均绝对误差(MAE)和Pearson相关系数(PCC)对预测器进行了优化和评估。我们应用了10倍交叉验证,并使用独立的测试数据集来可靠地估计我们模型的性能。构建模型的关键步骤是:
- 我们测量了在权重优化和没有优化的情况下预测器的性能。采用遗传算法进行优化。结果表明,优化后PCC提高0.96%,MAE提高6.14%。
- 我们将正则化回归算法的核值从1变到4,以找到训练器的最佳参数。3次多项式函数提供了最好的结果。
- ASA的最终预测因子REGAd的PCC和MAE值3.P值分别为0.7337和23.9%。
- 我们报告了案例研究来描述预测器的有用性。
- 我们的分析表明,预测的ASA值与氨基酸的物理性质非常一致。
- 最后,我们将预测的ASA应用于与蛋白质结构预测相关的关键应用。我们整合了ASA预测的结果来改进我们现有的能量函数3DIGARS的性能。我们将与ASA预测相关的误差转换为能量分量,并在基准诱饵测试集的基础上将我们开发的能量函数3DIGARS提高了32.32%。
出版
改进的可达表面积预测导致有效的能量函数应用。
Iqbal S, Mishra A, Hoque MT
《理论生物学》2015年9月7日










留言回复
你一定是登录发表评论。