进化实验中微进化驱动基因的自动识别
实验室里的科学家经常进行所谓的“进化实验”,他们把微生物放在它不习惯的环境中,等待它在基因层面上适应这种环境(进化)。由于这种“进化”发生得非常迅速,而且不会导致新物种的产生,而只是对微生物的功能进行少量的调整,因此这被称为微进化。
进化实验完成后,通过对进化微生物的基因组进行测序,收集微生物基因表达信息(RNA-seq或微阵列),生成数据。这导致了大量的数据,手工解释起来很麻烦。这就是为什么需要自动分析这些大数据集的算法来指导进一步的研究。官方manbetx手机版这种算法必须回答的一个重要问题是:在观察到的(有时是许多)突变中,哪些是适应新环境的原因?为了给这个问题提供一个充分的答案,我们开发了一个叫做“PheNetic”的算法。
为了回答这个问题,PheNetic使用了所研究微生物的“全基因组相互作用网络”。官方manbetx手机版这是一张微生物的蓝图,其中包含了其基因如何相互作用的信息。交互网络是必要的,因为通常一个进化实验要重复几次。每次,生物体的结果(表型)将非常相似,但导致这种结果的潜在突变可能不相同。虽然不相同,但这些突变往往发生在相同的生物学途径中,因此在全基因组相互作用网络中彼此接近。PheNetic通过在全基因组相互作用网络中寻找相互重叠的相关“路径”来利用这一特性。
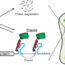
图1给出了PheNetic的概述:它使用基因的表达数据来重新权衡相互作用网络,产生一个概率子网络,其中两个基因之间的每个相互作用都有“机会”参与。然后,在突变基因与高度上下调控基因之间的相互作用网络上寻找路径。它为每次重复进化实验(焦点终点)执行这些步骤。在最后一步,使用概率框架,PheNetic整合所有焦点端点的所有可能路径,并选择一个包含所有焦点端点之间重叠程度最高的最可能路径的子网。
我们分析了两种非常不同的进化实验,使用PheNetic:获得抗生素耐药性(阿米卡星)大肠杆菌一个种群的进化大肠杆菌变成一个包含两种形态和功能截然不同的“生态型”的种群大肠杆菌它们可以共存。在阿米卡星耐药的情况下,我们能够自动识别涉及NADH脱氢酶和末端链氧化酶的几个基因,这两个过程都可以影响阿米卡星摄取所依赖的质子-基序力。在共存的生态型情况下,我们已经知道有两个特定的基因促成了共存(arcA三羧酸循环的一个组成部分现货(严格响应的一个组成部分)。PheNetic发现这两个基因对表现型都很重要,有趣的是,它还发现了另一个基因(acs),这是一种细胞外的醋酸清除剂,这意味着醋酸在一定程度上负责两种生态型之间的交叉摄食机制。
综上所述,“PheNetic”等算法对于大数据分析的可行性和速度至关重要。这在进行大规模实验以发现表现出低信号的重要遗传机制时尤其重要,因为如。它们很罕见。未来可以使用该技术分析的重要例子是大规模癌症数据集(数千名患者),以确定药物靶点的优先顺序,以及乙醇耐药性数据集,以提高生物乙醇的生产。
Bram Weytjens, Dries De maiyer, Kathleen Marchal
信息技术部(INTEC, iMINDS),根特9052,比利时
根特大学植物生物技术与生物信息系
科技园927,9052根特,比利时
比利时根特生物信息学研究所,根特科技园927,9052
鲁汶大学微生物与分子系统系,
比利时鲁汶B-3001 Kasteelpark Arenberg 20
出版
基于网络的eQTL数据分析以确定驱动突变的优先级。
张建军,张建军,李建军,等
2016年1月













留言回复
你一定是登录发表评论。