一种新的基于深度学习的预测rna -蛋白质相互作用的方法
rna结合蛋白(rna binding proteins, rbp)占真核生物蛋白质组的5-10%,调控基因的定位和翻译。另一方面,rbp的突变已被发现与疾病风险相关,如肌萎缩性侧索硬化症中的FUS和TDP-43。因此,解码rna和蛋白质之间的联系有助于深入了解其背后的机制。通过实验方法鉴定ncRNA相互作用仍然具有挑战性和高成本,这可以通过使用计算模型来补充。如何准确、自动地识别RNA是否与蛋白质结合是迫切需要的。

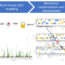
图1所示。将RNA和蛋白质序列编码为k-mer频率的载体。这20种氨基酸的分类如下:(Ala、Gly、Val)、(Ile、Leu、Phe、Pro)、(Tyr、Met、Thr、Ser)、(His、Asn、Gln、Tpr)、(Arg、Lys)、(Asp、Glu)和(Cys)。
我们开发了一种基于深度学习的方法,IPMiner,可以直接从序列中自动预测RNA-蛋白质相互作用,该方法可以应用于任何RNA和蛋白质对。新的IPMiner执行以下4个步骤:
在IPMiner的第一步(图1)中,它编码RNA和蛋白质序列的简单k-mer序列特征。对于RNA序列,我们提取4-mer的频率,即4-mer在序列中出现的次数。对于蛋白质序列,我们首先将20个氨基酸分成7组,然后使用还原的氨基酸字母表得到3-mers的频率。
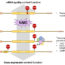
在步骤2中,我们使用堆叠式自编码器分别进一步细化蛋白质和rna的原始k-mer特征的表示(图2)。堆叠式自编码器由多层神经网络组成,每层经过非线性变换后重建原始输入。
在步骤3中,将从堆叠的自编码器中学习到的蛋白质和rna的高级特征连接起来,并将其馈送到随机森林分类器中以预测该rna -蛋白质对是否相互作用。为了消除单个分类器造成的潜在偏差并提高准确率,我们还训练了另外2个随机森林分类器:一个是使用未经任何后处理的原始k-mer频率特征作为输入,另一个是使用未微调的无监督堆叠自编码器抽象特征,使用标记rna -蛋白质对作为输入。总的来说,我们将有3个随机森林分类器用于不同的输入特征,作为彼此的补充。
在步骤4中,最后我们使用堆叠集成来集成这3个不同分类器的输出,其中来自3个不同分类器的输出被输入到逻辑回归中,以学习3个不同分类器的权重。与传统的多数投票相比,它可以自动学习不同分类器对最终决策的不同贡献。
由于新的IPMiner只需要序列作为输入,它可以用来预测任何一对rna和蛋白质相互作用的概率。其有效性已在多个rna -蛋白数据集上得到证实。为了使我们的IPMiner更好地服务于学术界,一个易于使用的独立软件已经在http://www.csbio.sjtu.edu.cn/bioinf/IPMiner/和https://github.com/xypan1232/IPMiner上发布。使用IPMiner时,用户只需要分别为rna和蛋白质准备两个Fasta文件,IPMiner就会自动计算两个文件中任意一对rna和蛋白质之间的相互作用电位。
小勇锅1,hong bin沈2
1荷兰鹿特丹伊拉斯谟医学中心医学信息部
2上海交通大学图像处理与模式识别研究所
系统控制与信息处理教育部重点实验室
出版
IPMiner:隐藏的nrna -蛋白质相互作用序列模式挖掘与堆叠自编码器准确的计算预测。
潘鑫,范云云,严军,沈海鹏
BMC Genomics, 2016年8月9日











留言回复
你一定是登录发表评论。